近日,亿纳谱团队联合上海交通大学、上海交通大学医学院附属仁济医院、中山大学附属中山纪念医院在eBioMedicine (IF=10.8)上发表题为“Serum metabolic fingerprints encode functional biomarkers for ovarian cancer diagnosis: a large-scale cohort study”的研究论文,该研究提出使用纳米粒子增强激光解吸电离质谱(NELDI-MS)快速获取血清代谢指纹(SMFs),结合机器学习挖掘代谢标志物,旨在突破现有技术瓶颈,提升卵巢癌(OC)早期诊断的准确性和临床适用性。
研究背景
卵巢癌(OC)是致死率最高的妇科恶性肿瘤,早期诊断至关重要但充满挑战。现有诊断方法(如经阴道超声和血清生物标志物CA-125、HE-4)的敏感性和特异性有限 。代谢组学为发现新的诊断生物标志物提供了潜力,但之前的研究受限于样本量较小、分析技术传统和缺乏生物学功能验证 。
关键科学问题
是否可以利用大规模人群队列的血清代谢指纹图谱(SMFs),通过先进的质谱技术和机器学习方法,识别并验证一组可靠的、具有临床应用潜力的非侵入性生物标志物,以提高卵巢癌(特别是区分恶性与良性卵巢肿块)的诊断准确性?
技术路线
首先构建了一个包含 1432 名受试者的大规模 OC 相关队列,包括 662 名 OC、563 名良性卵巢疾病和 207 名健康对照受试者,涵盖回顾性 (n = 1073) 和搁置验证 (n = 359) 队列。使用纳米颗粒增强激光解吸/电离质谱 (NELDI-MS) 记录血清代谢指纹 (SMF)。通过对发现队列中的 SMF 进行机器学习开发了一个诊断面板,并在独立验证和预留验证队列中进行了验证。使用液相色谱MS进一步验证已鉴定的代谢生物标志物,并在OC细胞系中评估其生物学功能。
创新点
构建了迄今为止用于血清代谢生物标志物发现和验证的最大规模OC相关队列 。应用了先进的NELDI-MS技术,实现了快速(约30秒/样本)、低成本(约2-3美元/样本)的SMFs分析,适用于大规模临床应用 。识别并验证了一个具有良好诊断性能的四代谢物生物标志物组合,并证明其与ROMA结合能进一步提高诊断效能 。对发现的生物标志物进行了初步的生物学功能验证 。
研究结果
1. 队列特征和研究设计
本研究建立卵巢疾病生物库,收集1691例血清样本,剔除术前放化疗、转移性肿瘤等特殊病例后,纳入662例OC和770例非OC受试者(含563例良性和207例健康对照)进行分析。其中1073例(OC 419例、良性447例、健康对照207例)用于回顾性队列,359例(OC 243例、良性116例)用于验证队列(图1a)。
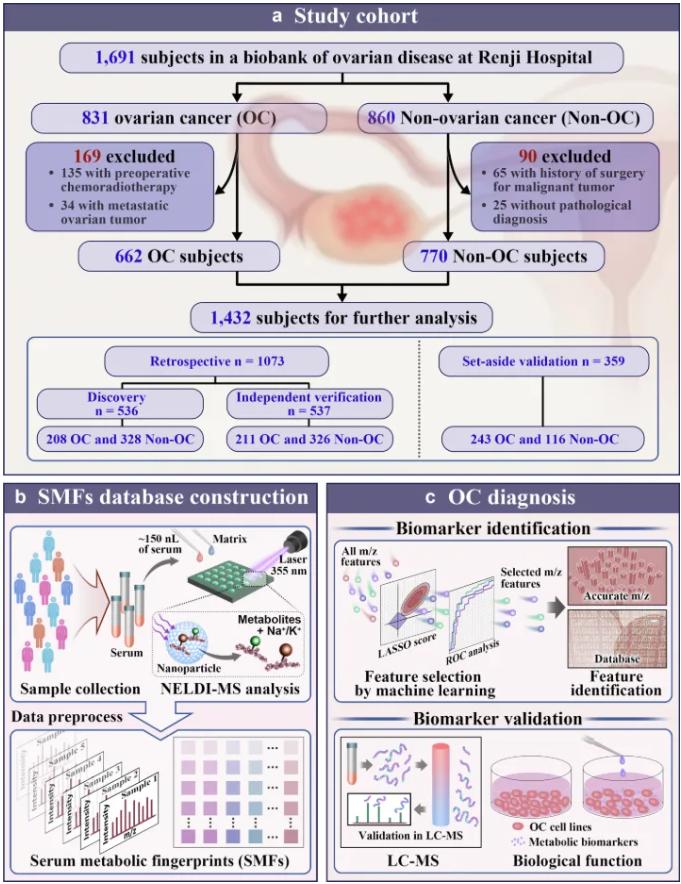
研究设计
首先,利用NELDI-MS构建OC相关SMFs数据库;在NELDI-MS中,在355 nm激光(Nd:YAG激光)下用纳米颗粒基质分析150 nL血清样品,记录原始质谱,并对其进行预处理以获得SMF(图1b)。然后,通过机器学习SMFs的特征选择和数据库中的精确m/z搜索,确定了代谢生物标志物组,包括葡萄糖、组氨酸、PCA和二氢胸腺嘧啶(图1c)。将这些代谢生物标志物与ROMA(一种结合CA-125,HE-4和绝经状态的风险评估算法)相结合,实现了OC的诊断性能。最后,使用LC-MS验证了4种代谢生物标志物,并评估了它们对体外OC细胞生物学行为的影响,包括增殖,集落形成,迁移和细胞凋亡(图1c)。
2. OC关联SMFs数据库的构建
癌细胞代谢重编程导致的癌症患者血液中的代谢改变已被广泛报道。这些发现表明,基于血液的代谢分析可以作为癌症的可靠指标。首先使用NELDI-MS构建了回顾性队列的OC相关SMFs数据库,展示了分析速度快、通量高和重现性理想的优点。
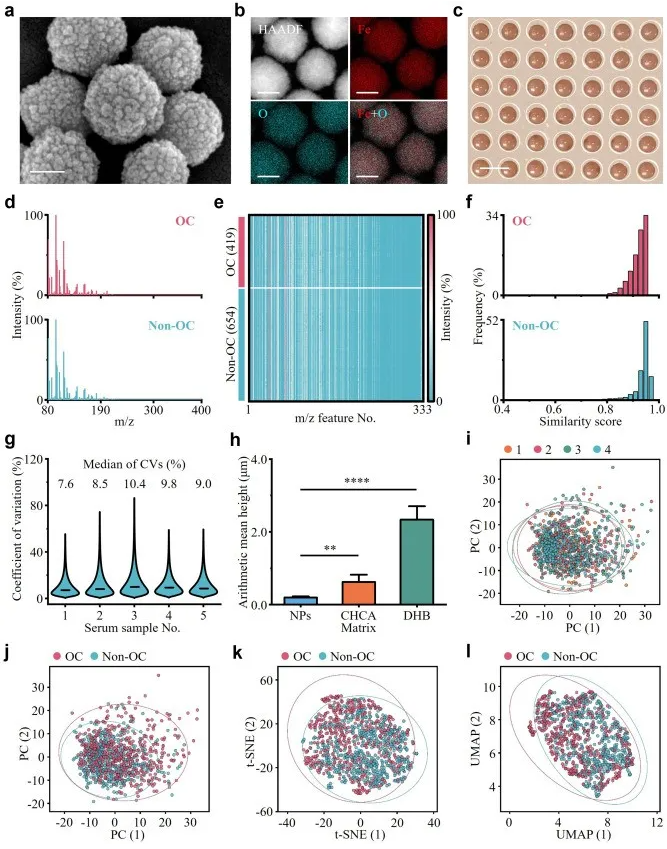
图2 OC相关SMFs数据库的构建。
但是在上述数据可视化中,SMF的无监督分析显示OC组和非OC组重叠(图2j-l),表明有必要引入监督机器学习算法来解码血清代谢指纹(SMF)以进行OC诊断。
3. 用于OC诊断的SMF的机器学习
采用机器学习进行模型构建,以研究 SMF 在区分 OC 和非 OC 群体方面的潜力。在发现和独立验证队列中,PRC 下的面积分别为 0.88-0.90 和 0.88-0.89(图 3f 和 g)。PRC 分析结果与 ROC 分析一致,证实 SMFs 在 OC 诊断方面表现出理想的性能。
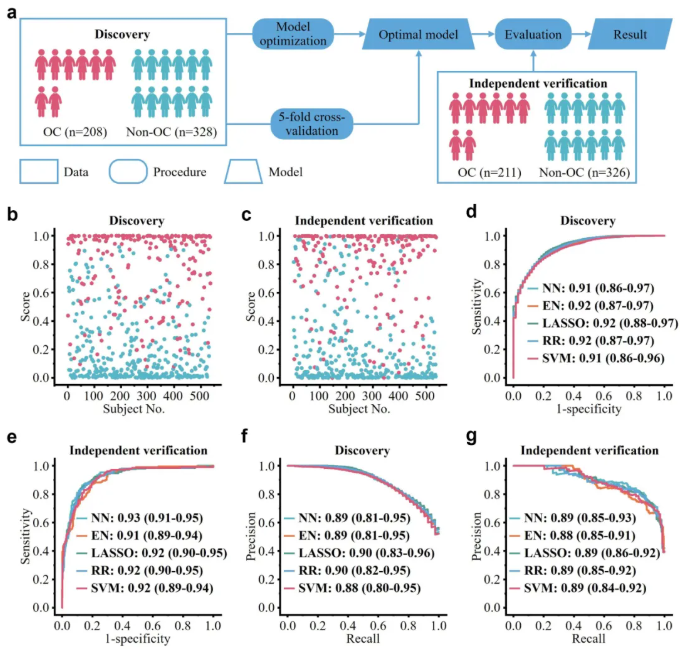
图 3 用于 OC 诊断的 SMF 机器学习。
4. 代谢生物标志物面板的鉴定和验证
为了促进 SMFs 在大规模临床环境中用于 OC 诊断的实用性,研究人员仅通过使用发现队列的系统特征选择确定了稀疏且可靠的代谢生物标志物面板, 获得由 4 种代谢物组成的代谢生物标志物组。
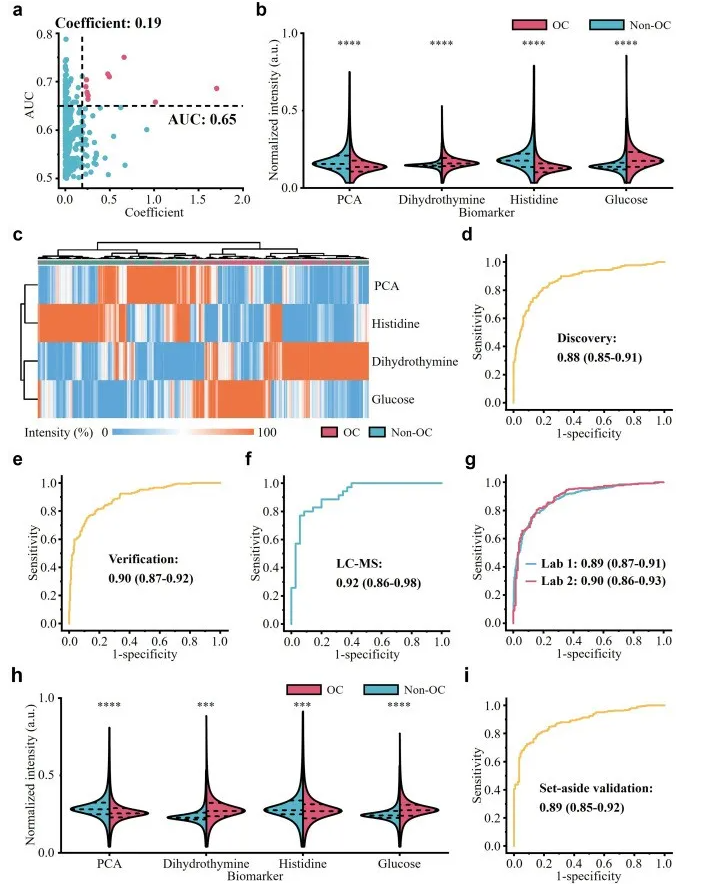
图 4 代谢生物标志物面板的鉴定和验证。
4 种代谢生物标志物的聚类分析区分了 ∼72% OC 和非 OC 受试者,表明已识别的生物标志物组具有诊断潜力(图 4c)。
5. 与现有诊断生物标志物的性能比较
在发现和独立验证队列中比较了代谢生物标志物与现有 CA-125、HE-4 和 ROMA 生物标志物的诊断性能。代谢生物标志物组显示AUC为0.89(95%CI:0.84–0.94),与ROMA(AUC = 0.88,95%CI:0.82–0.93)相当,高于CA-125(AUC = 0.86,95%CI:0.80–0.92)和HE-4(AUC = 0.85,95%CI:0.78–0.91)用于OC诊断(图5a)。
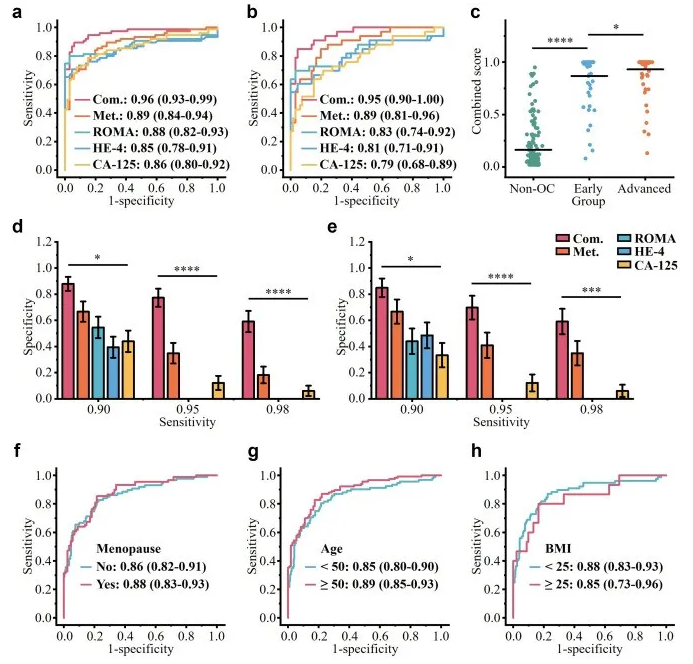
图5 与现有诊断生物标志物的性能比较。
6. 代谢生物标志物的生物学功能验证
采用了 2 种代表性的 OC 细胞系 HEY 和 OVCAR-8 来评估代谢物的生物学功能,包括细胞增殖、集落形成、迁移和凋亡。
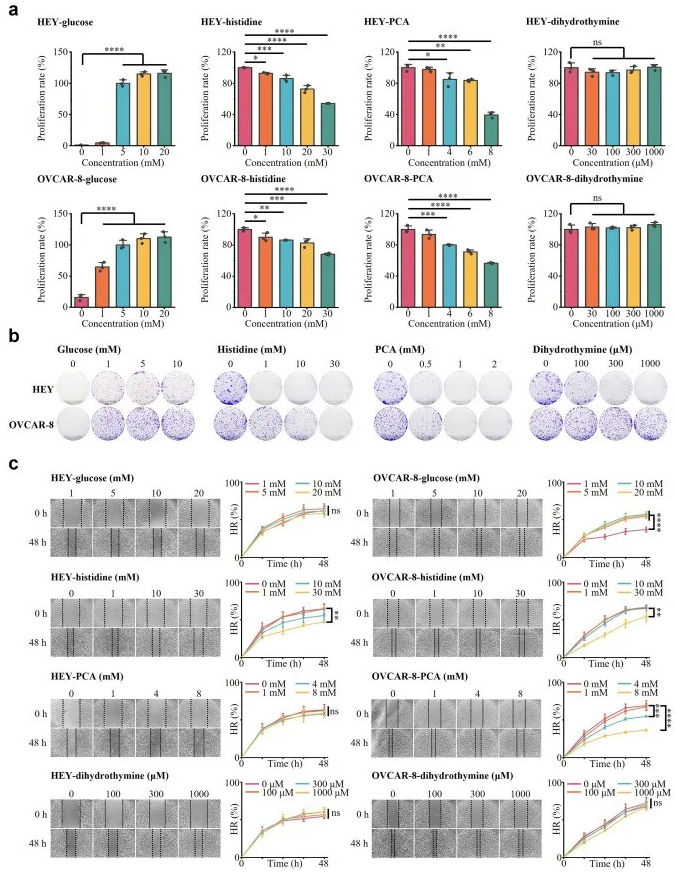
图6 代谢生物标志物的生物学功能验证。
局限性与不足
研究依赖特定的质谱仪和纳米粒子技术,其临床转化可能需要进一步的工程改造以适应即时检测(Point-of-care testing)的需求 。虽然包含了独立的验证队列,但仍需在更多中心进行外部验证以确认结果的普适性 。生物学功能验证是初步的,需要更复杂的模型(如类器官和动物模型)来深入探究这些代谢物在OC发病机制中的具体作用 。

